| 일 | 월 | 화 | 수 | 목 | 금 | 토 |
|---|---|---|---|---|---|---|
| 1 | 2 | 3 | 4 | 5 | 6 | 7 |
| 8 | 9 | 10 | 11 | 12 | 13 | 14 |
| 15 | 16 | 17 | 18 | 19 | 20 | 21 |
| 22 | 23 | 24 | 25 | 26 | 27 | 28 |
| 29 | 30 | 31 |
- 파이토치
- 파이썬
- Python
- opencv
- Semantic Segmentation
- 논문
- Paper Review
- programmers
- 논문 리뷰
- 논문리뷰
- Segmentation
- pytorch
- 코드구현
- 인공지능
- Convolution
- 머신러닝
- 딥러닝
- transformer
- 알고리즘
- 코딩테스트
- optimizer
- Ai
- 프로그래머스
- Self-supervised
- cnn
- 옵티마이저
- object detection
- Computer Vision
- 논문구현
- ViT
- Today
- Total
Attention please
[논문 리뷰] U-Net: Convolutional Networks for BiomedicalImage Segmentation(2015) 본문
[논문 리뷰] U-Net: Convolutional Networks for BiomedicalImage Segmentation(2015)
Seongmin.C 2023. 7. 2. 20:25이번에 리뷰할 논문은 U-Net: Convolutional Networks for BiomedicalImage Segmentation 입니다.
https://paperswithcode.com/paper/u-net-convolutional-networks-for-biomedical
Papers with Code - U-Net: Convolutional Networks for Biomedical Image Segmentation
🏆 SOTA for Semantic Segmentation on Kvasir-Instrument (DSC metric)
paperswithcode.com
2015년도에 나온 논문이며 Image segmentation 분야에서 아주 큰 영향을 준 논문이라고 해도 과원이 아닙니다. UNet 모델에서 파생된 논문들도 다양하니 후에 리뷰하는 시간을 가져보도록 하겠습니다.
Abstact
deep network를 성공적으로 학습하기 위해서는 수천개의 annotation이 달린 training data가 필요합니다. 본 논문에서는 보다 더 효율적으로 사용가능한 annotation을 사용하기 위해 강한 data augmentation 사용에 의존하는 훈련 전략을 세웁니다. 저자가 제시하는 모델 architecture는 context capture를 위한 contracting path와 대칭적으로 확장되는 expanding path로 구성됩니다.
본 논문에서는 아주 적은 양의 데이터로 제안된 network를 end to end 로 학습하였으며 ISBI challenge 에서 이전의 sliding-window convolutional network 와 같은 이전의 best method를 능가함을 보여주었습니다. 또한 ISBI cell tracking challenge 2015 에서도 같은 network를 사용하여 큰 차이를 두고 우승을 했습니다.
또 이 network는 빠르다는 장점이 있습니다. 2015년의 GPU 성능으로 512x512 이미지를 segmentation 하는데 1초도 걸리지 않는다고 합니다.
Introduction
일반적으로 convolutional network은 classification task에서 사용됩니다. 이미지의 output이 1개의 class label로 출력되는 형식이죠. 하지만 많은 visual task 그중에서도 biomedical image인 경우 output이 위치 정보를 포함하여야 합니다. 즉, 각각의 pixel에 class label이 지정되어야 한다는 것입니다. 게다가 Deep learning network를 성공적으로 학습시키기 위해 필요한 것들 중 하나인 수천개의 데이터들을 biomedical task에서 구하는 것은 불가능에 가깝습니다.

이런 점들을 보완하여 나온 Network in a sliding-window setup 모델이 존재합니다. 이는 각 pixel에 대해 class를 예측하기에 localize하다고 할 수 있습니다. 또한 patch에 대해 학습을 진행하기 때문에 기존의 training image들보다 더 큰 양으로 가지고 학습을 합니다. 이 모델은 ISBI 2012 대회에 큰 차이로 우승을 하기도 했죠.
하지만 위 전략은 두가지의 단점을 가집니다.

첫번째는 각각의 patch에 대해 개별적으로 학습을 진행하며, 겹치는 patch들 때문에 많은 중복이 있기 때문에 속도가 느리다는 것이며, 두번째는 localization 정확도와 context 사용이 trade-off 관계를 가진다는 것입니다.
본 논문에서는 기존의 FCN(fully convolutional network) 를 더 우아한 네트워크로 만들었다고 합니다.
본 논문에서 소개하는 모델의 main idea는 pooling 연산자가 upsampling 연산자로 대체되는 연속적인 layer를 통해 일반적인 축소 network를 보완하는 것입니다. upsampling 연산자를 가지기 때문에 이 layer들은 output의 해상도를 증가시키죠. 또한 localize를 위해 contracting path는 upsampling된 output과 결합됩니다.

해당 architecture의 한가지 중요한 수정사항은 upsampling을 하는 부분에도 많은 수의 feature channel들을 가진다는 것입니다. 이는 더 높은 해상도의 layer에 context information을 전송할 수 있도록 도와줍니다. 결과적으로 expansive path는 contracting path와 거의 비슷하게 대칭적이며, 이 모양이 마치 U자와 같다 하여 이 모델의 이름을 UNet이라 부르죠.
UNet은 fc layer이 존재하지 않으며 오직 convolution 연산만으로 이루어져있습니다. 또한 segmentation map은 input image의 context 정보만을 가지는 pixel들만을 포함한 형태가 됩니다. segmentation task에서 context information은 영역이 되기 때문에 찾고자 하는 object의 영역 정보를 output으로 출력한다는 의미가 됩니다.
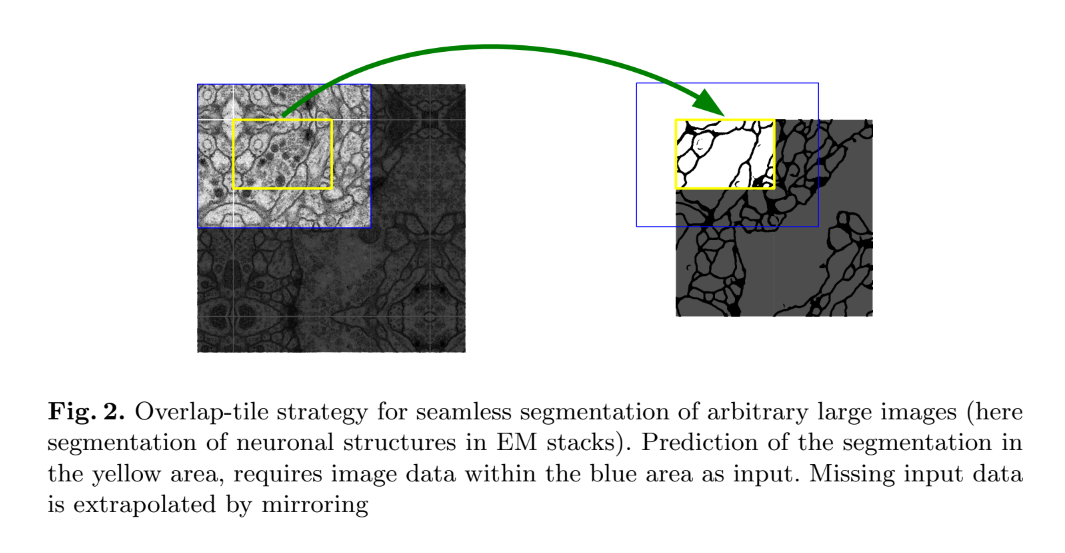
또한 본 논문에서는 Overlap-tile 전략을 소개하는데 이는 큰 이미지에 대해 매끄러운 segmentation이 가능하도록 하기 위한 전략입니다. 위 figure에서 영역을 분할하고자 하는 영역은 노란색 영역이며, 이 영역을 분할하기 위해 파란색 영역을 사용합니다. 또한 이미지의 가장자리 에 대한 pixel을 예측하기 위해서 input image를 mirroring 하여 채워나갑니다.
위 그림으로 예시를 들자면 오른쪽 그림의 파란색 영역을 살펴보면 빠져있는 부분이 존재합니다. 이 부분을 채우기 위해 대칭이 되도록 image를 채워넣기 때문에 왼쪽 그림과 같이 한층 더 해상도가 커져있는 상태가 됩니다. 이렇게 mirroring을 하는 이유는 본 데이터가 세포라는 특성 때문인데 보통 세포는 대칭 구조를 지니기 때문에 대칭되게 하여 padding을 하였다고 합니다. (보통 가장자리를 학습하기 위해서 zero padding을 사용합니다.)
또한 의료 분야에서 사용할 수 있는 데이터의 양은 아주 제한적입니다. 본 논문에서는 이를 위해 탄성 변형(elastic deformations) 를 사용하여 data augmentation을 적용했습니다. 대칭적으로 padding을 채우는 것 역시 탄성 변형의 한 예라고 볼 수 있죠.

cell segmentation task에 또 한가지의 challenge가 남아있습니다. 위 figure를 보면 세포들이 서로 접해있는 부분이 많음을 확인할 수 있는데 이와 같이 같은 클래스를 가지면서 서로 닿아 있는 객체들을 분리해야합니다. 본 논문에서는 이 문제를 해결하기 위해 weighted loss를 사용했는데 서로 닿아 있는 cell 사이에 background label을 사용하여 분리한 후 이 background label에 대해 loss function에 큰 가중치를 얻게 합니다.
Network Architecture
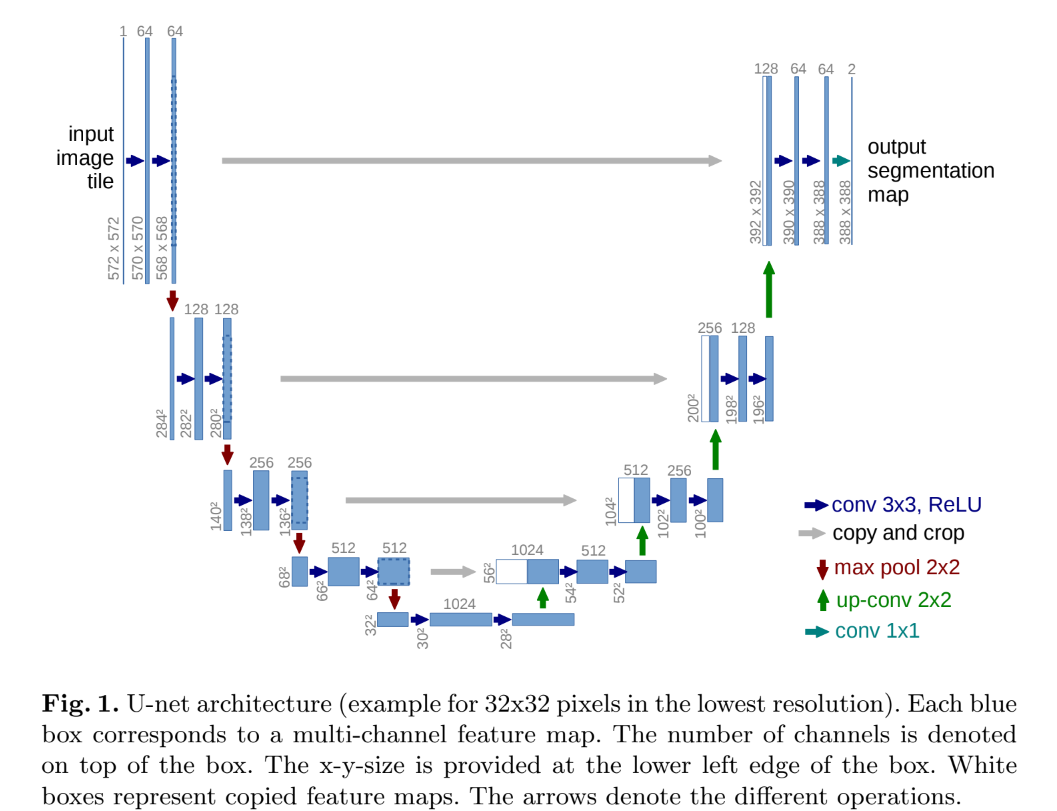
위 figure는 전체적인 U-Net의 Architecture를 보여줍니다. 왼쪽 부분에 해당하는 부분은 contracting path이며, 오른쪽에 해당하는 부분은 expansive path 입니다. contracting path는 일반적인 convolution network의 구조와 동일하게 진행됩니다. 2개의 3x3 convolution, ReLU, 2x2 max pooling (2 stride) 가 반복되어 진행됩니다. 각 downsampling step 에서는 feature channel의 수가 2배가 되죠.
이제 upsampling의 차례입니다. expansive path의 모든 step은 feature map을 upsampling을 한 후 2x2 convolution을 사용하여 feature channel의 개수를 절반으로 줄입니다. 그 후에 contracting path에서 크기가 동일하도록 crop한 feature map을 concatenation을 한 후 2개의 3x3 convolution, ReLU가 적용되게 됩니다. 밑 그림을 보면 알 수 있듯이 concatenation되는 두 feature map의 해상도가 다릅니다.

contracting path에 해당하는 feature map의 경우 size가 64x64 인데 expansive path에 해당하는 feature map의 크기는 56x56 인 것을 확인할 수 있습니다. 이와 같이 서로 크기가 다른 map을 concate하는 것은 불가능하기 때문에 crop을 하여 크기를 맞추어준 후 concatenation을 진행합니다.
또한 convolution과 pooling layer를 반복하여 계산하게 되면 이미지의 크기가 줄어들며 2x2 size의 max pooling 연산은 image의 height 와 weight를 반으로 줄입니다. 그렇기 때문에 input image의 크기가 홀수인 경우 pooling 연산을 통해 output image의 크기가 홀수가 되기 때문에 정확한 연산이 불가능합니다. 이 문제를 해결하기 위해 input image의 크기는 짝수로 설정해야합니다.
Training
UNet 모델은 padding이 존재하지 않는 convolution 연산으로 이루어져있기 때문에 output image는 input image에 보다 일정한 폭만큼 줄어들게 됩니다. GPU 메모리를 최대한으로 활용하고, overhead를 최소화하기 위해 큰 batch size 보다는 큰 크기의 input tile을 선호합니다. 이런 이유로 batch size를 한 이미지로 줄이게 되죠. 또한 높은 momentum (0.99) 을 사용하여 이전에 본 train sample의 대부분이 현재 최적화 단계의 업데이트를 결정하게 됩니다.
UNet 모델의 연산 과정을 모두 거친 후 나온 feature map에 대해서 pixel 별로 softmax를 적용한 후 loss function 인 cross entropy를 적용합니다.

위 식은 softmax에 대한 식입니다. K는 pixel의 개수이며 각각의 pixel에 대해 확률값을 도출합니다.
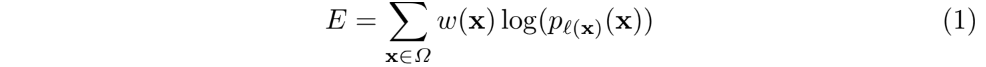
위에서 구한 각 pixel들의 softmax 값들을 가지고 loss function인 cross entropy 값을 계산합니다. 또한 추가로 가중치 w(x) 가 곱해진 것을 확인할 수 있는데 이는 일부 pixel에 대해 더 많은 중요성을 부여하기 위해 곱해집니다. 그렇다면 이 가중치 w(x) 는 어떻게 구성될까요?
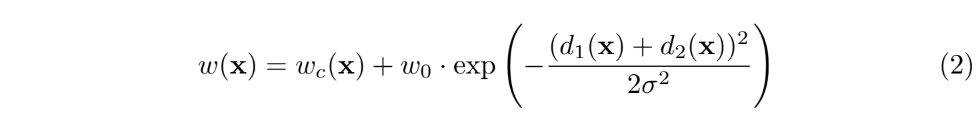
먼저 weight map은 실제 segmentation이 대해 사전에 계산됩니다. train dataset에서 특정 class의 pixel 빈도의 차이를 보완해주며, 서로 닿는 cell 사이에 구분을 위해 도입한 작은 분리 경계에 대해 학습을 강제합니다. wc 의 경우 class의 빈도를 균형잡기 위한 weight map이며, d1은 가장 가까운 cell의 경계까지의 거리를 나타내며, d2는 두 번째로 가까운 cell의 경계까지의 거리를 나타냅니다. 본 논문에서는 w0의 값으로는 10으로, $ \sigma $값으로는 5pixel로 설정하였습니다.
Experiments
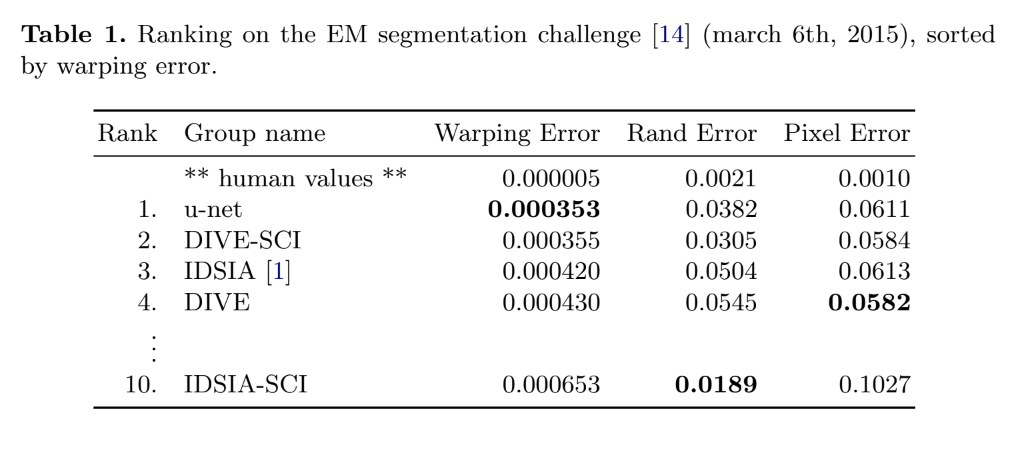
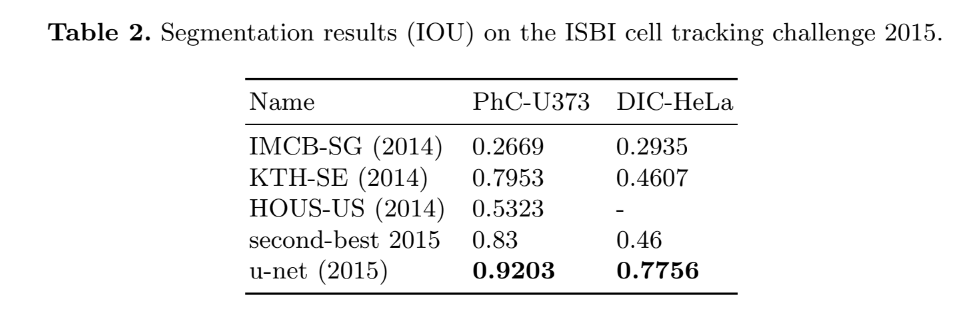
위의 Table 1, Table 2를 보면 UNet의 성능이 다른 모델에 비해 좋은 성능을 보여주는 것을 확인할 수 있습니다.
Conclusion
UNet 모델은 다양한 biomedical segmentation 분야에서 좋은 성능을 보여줍니다. 또한 elastic deformation을 통해 Data augmentation을 적용하여 적은 양의 데이터만으로도 좋은 성능을 보여주었으며 GPU(6 GB) 에서 10시간만에 훈련이 완료될 정도로 빠른 훈련 속도를 지니고 있습니다.




